微生物Meta测序
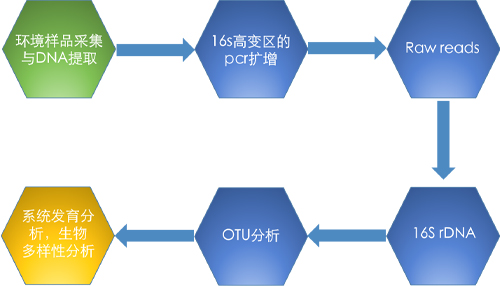
16S rDNA序列(在所有物种中高度保守)是最常用的细菌分类标准,通过提取环境样品的DNA,并扩增其中16S rDNA基因;通过检测16S rDNA 序列的变异和丰度,反映环境样本中细菌的分类和丰度。16S rDNA 有9个高变区,V6,V3,V4 区可以用来鉴定绝大多数细菌,基于Illumina HiSeq 2000平台的16S rDNA测序可以将V6,V3,V4区正反向读通,提供环境样本物种分类,物种丰度,种群结构,系统进化,群落比较等诸多信息,在微生物分类鉴定,微生态研究方等面起到重要作用。
应用领域
环境微生物分类、丰度分析
环境微生物群体基因集构建
分析内容
1. 标准信息分析
去除低质量,污染,引物序列,将PE reads拼接成Tag 等。
2. 高级信息分析
a) 对unique tags 聚OTU,并进行物种注释
b) 单样品复杂度的Alpha diversity分析
c) 物种(或OTU)丰度分析
客户需提供的样品:
样品类型:PCR扩增产物;
样品需求量:小片段文库,≥ 4 μg(浓度≥30 ng/μl;纯度OD260/280=1.8~2.0);
总样品量需根据实验策略,如建库类型及建库数量而定;
项目周期
根据不同样品数和送样时间不同周期不同,一般约为45个工作日。
完成标准:对每个样品,产生不低于合同规定的Clean tags数量和Clean data数据量。