Cytoscape是一款用于分析和图形化展示网络的软件。Cytoscape最基本的组织形式是网络图,分子(基因或者蛋白)表示一个节点,分子间的相互作用表示一个边。可以用来对提供的网络结构数据进行可视化分析。
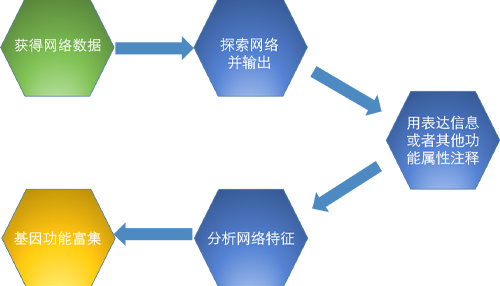
二、方法
1. 获得网络数据(Obtain network data);
2. 探索网络并输出(Explore network and generate layout);
3. 用表达信息或者其他功能属性注释(Annotate with attribute and expression data);
4. 分析网络特征(Analyze network features);
5. 基因功能富集(Detect enriched gene functions)。
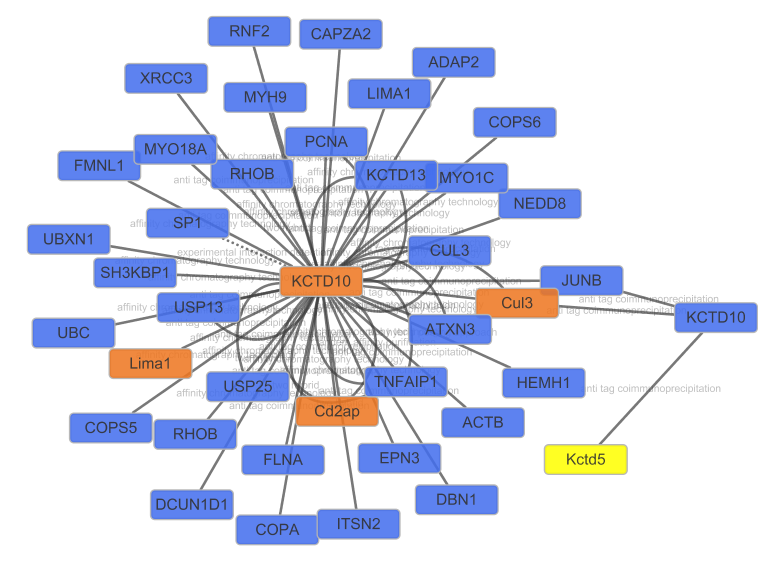
需要提供的数据资料:可提供以下几种形式的数据
1.所要探究的基因列表
2..sif(相互作用)文件(.sif的每一行包含三个标识,第一个标识是源节点,第二个标识是相互作用类型,第三个标识是目标节点。)
3..noa(节点文件)(第一行是属性名,例如’SubcellularLocation’,接下来的每一行包含一个节点名、等号、节点属性值,属性值可以是数值型也可以是字符型,例如’4.12’或者’nucleus’。)
4..eda(边文件)(边文件跟节点文件类似,这里的边名由源节点、相互作用类型、目标节点组成。)
5. 表达值文件。(表达值数据是一个矩阵,每一行代表一个基因或者蛋白在不同实验条件下的表达值。第一行是列标签,第一列是基因名,第二列是基因的描述,接下来的列是表达值,每一个实验一列。如果数据包含P-value或者其他衡量显著性的值,每一个实验则对应两列,第一列表达值,第二列P-value,这两列的列标签必须相同。)
返还结果:CytoScape可视化图,若有需要可进一步性寻找关键基因等相关分析。