GO注释
一、GO(gene ontology)
GO(gene ontology)是基因本体联合会(Gene Onotology Consortium)所建立的数据库,旨在建立一个适用于各种物种的,堆积因和蛋白质功能进行限定和描述的,并能随着研究不断深入而更新的语言词汇标准.GO是多种生物本体语言中的一种,提供了三层结构的系统定义方式,用于描述基因产物的功能。
二、GO的组成
1.基因执行的分子功能(Molecular Function)
2.基因所处的细胞组分(Cellular Component)
3.基因以及参与的生物学过程(Biological Process)
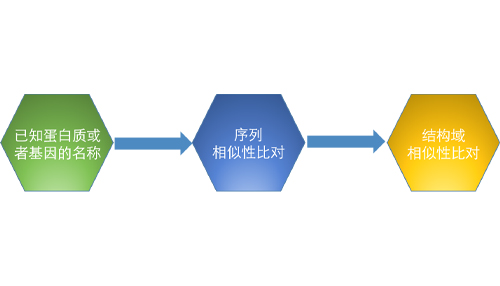
三、GO注释方法
(1)序列相似性比对(BLAST)
(2)结构域相似性比对(InterProScan)
需要的文件:所关注的蛋白质或者基因的名称或者ID。
返还结果:对应功能注释图,对于提供基因列表的数据可以提供具有参考意义的hub基因。
KEGG注释
一、KEGG
KEGG(Kyoto Encyclopedia of Genes and Genomes)是系统分析基因功能、基因组信息数据库,它有助于研究者把基因及表达信息作为一个整体网络进行研究。KEGG现在由6个各自独立的数据库组成,分别是基因数据库(GENES database)、通路数据库(PATHWAY database)、配体化学反应数据库(NGAND database)、序列相似性数据库(SSDB)、基因表达数据库(EXPRESSION)、蛋白分子相互关系数据库(BRITE)等。KEGG是进行生物体内代谢分析、代谢网络研究的强有力工具。
二、KEGG的组成
1.基因数据库(GENES database) 含有所有已知的完整的基因组和不完整的基因组。
2.通路数据库(PATHWAY database) 储存了基因功能的相关信息,通过图形来表示细胞内的生物学过程。
(1)代谢通路
(2)Ortholog group图表
(3)蛋白—蛋白反应
3.配体数据库(LIGAND database) 包括了细胞内的化学复合物,酶分子和酶反应的信息。
需要的文件:所关注的蛋白质或者基因的名称或者ID。
返还结果:对应基因功能注释图,如有需要可以进一步分析提供功能富集图及具有参考意义的hub基因。
基因富集分析(gene set enrichment analysis)
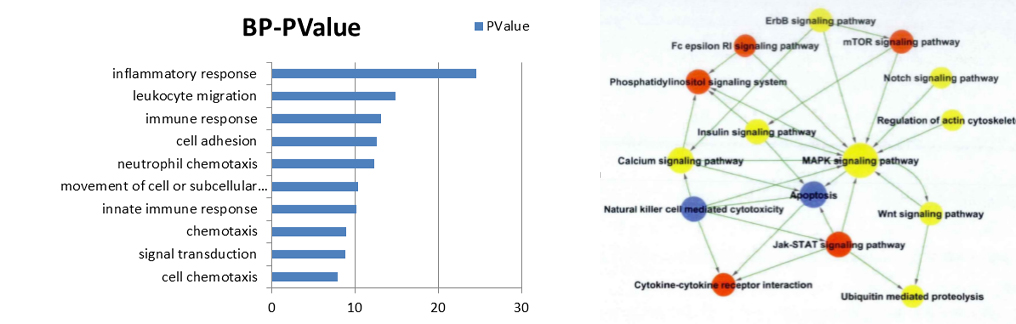
一、基因富集分析
基因富集分析(gene set enrichment analysis)是在一组基因或蛋白中找到一类过表达的基因或蛋白。一般是高通量实验,如基因芯片,RNA-Seq,蛋白质组学(质谱结果)的后续步骤。
基因富集分析需要我们提供某一类功能基因的集合用于背景,常用的注释数据库如:
The Gene Ontology Consortium: 描述基因的层级关系
Kyoto Encyclopedia of Genes and Genomes: 提供了pathway的数据库。
二、分析方法
1.GSEA:
1)GSEA是对全基因组表达谱芯片数据分析工具,根据已有的对基因的定位、性质、功能、生物学意义等知识的基础上,首先构建了一个分子标签数据库,数据库中包含了多个功能基因集。通过分析一组处于两个生物学状态的基因表达谱杂交数据,它们在特定的功能基因集中的表达状况,以及这种表达状况是否存在某种统计学显著性。GSEA是从另一个角度来诠释生物信息,可进一步完善我们对相关生物学事件的认识。
2)有如下特点:
计算所有输入基因集合的分数,而不是单个基因
不需要设置cutoff
找到一组相关的基因
提供了更加稳健的统计框架
需要提供的数据:分析的芯片表达谱数据。
返还结果:GSEA功能富集图。
2.DAVID
1) DAVID是一个生物信息数据库,整合了生物学数据和分析工具,为大规模的基因或蛋白列表(成百上千个基因ID或者蛋白ID列表)提供系统综合的生物功能注释信息,帮助用户从中提取生物学信息。
2)包含如下功能:
Gene Name Batch Viewer
Gene Functional Classification
Functional Annotation
Functional Annotation Chart
Functional Annotation Clustering
Functional Annotation Table
Gene ID Conversion
需要提供的数据:所关注的基因列表。
返还结果:基因列表的功能注释,基因名称转换,经过相应P-value及count数筛选过后的功能富集文件,及GO-BP,GO-CC,GO-MF,KEGG-Pathway功能富集图。